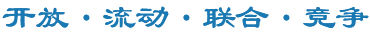| 代表性研究成果名称 | 类别 | 成果为第一完成单位 | 本室固定人员参加名单 | 所属研究方向 |
|---|---|---|---|---|
| 肺癌复发转移、耐药及预后判断系列研究 | 应用基础类 | 是 | 何建行、蒋义国、张必良、吕嘉春、李时悦、刘金保、梁文华 | 肺癌诊治 |
(一)研究背景
肺癌是人类最常见的恶性肿瘤之一,在全球及我国,其发病率和死亡率均位居所有癌症的首位。而且我国存在促进肺癌发生及发展的高危因素,不仅面临着3亿烟民以及被动吸烟群体的挑战,还受到日益严重的大气污染(如PM2.5)的威胁,肺癌患病率仍在以极高的速度持续增加。尽管近年来肺癌的临床治疗进展迅速,然而其5年总体生存率仍低于20%,面对庞大的暴露人群,目前肺癌的防治水平仍亟待提高。
肺癌是涉及多种基因突变的疾病群,中国患者肺癌基因突变的分布及位点不清,其基因突变图谱与西方人有差异,同时,部分肺癌患者病变转移较快,其机制不明。阐明这些规律,对于国人肺癌的发病及其转移的精准治疗极为重要。此外,既往在TNM分期指导下诊断的早期肺癌,经根治性手术治疗后仍有相当部分患者复发,为此进行的不加选择的巩固性化疗容易造成过度治疗,浪费资源,且损害健康。因而对手术后的预后的精确评估对随后的处理具有重要临床意义。
近年科学界致力于肺癌的精准防治研究,希望通过揭示肺癌关联的生物暴露标志、效应标志和易感标志物来实现肺癌高危人群的精准定位、早期诊断和精确诊疗,从而降低肺癌的危害。将肺癌阻断在早期是治愈肺癌的唯一希望,通过手术切除及术后降低复发的干预是关键环节。因此过去5年,肺癌学组聚焦肺癌精准医学领域,紧密围绕早期肺癌预防、诊断以及治疗三个关键环节,建设完成大型临床数据库及生物资源库,并通过一系列研究揭示了我国肺癌发病的主要环境危险因素和遗传易感分子,探索了肺癌复发转移相关的机制和治疗靶点,发掘出一批可用于肺癌诊断及预后评估的分子标志物并转化为临床工具,研制出多个新型的治疗药物,并初步探索了肺癌的细胞免疫治疗,极大促进了我国肺癌预防和临床实践的研究进展,为肺癌精准防治提供了科学依据。
(二)创新性成果
(1)绘制了亚洲人肺腺癌基因突变图谱及肺癌转录组表达谱
基因变异是肺癌发生发展的根本原因或关键环节,也是治疗的靶点。近年来肺癌尤其肺腺癌驱动基因改变的陆续发现,以及相对应靶向治疗的发展,极大地改善了该类患者的生存率。此前国际上仅有来自TCGA的欧美人群的肺癌变异图谱公布,对于肺腺癌患病率更高发的亚洲人群尚缺乏相关数据。本实验室对来自中国的335个原发肺腺癌和35个对应淋巴结转移瘤患者样本进行了高通量全基因组和转录组分析,首次描绘了亚洲人肺腺癌致病基因突变全景图,发现了亚洲人肺腺癌特有的基因变异标签以及多个既往未被报道的主要驱动基因变异(RHPN2, GLI3, MRC2);在来自转移瘤患者的肿瘤中显著富集了TP53突变,一些调控细胞骨架重塑过程的基因如RAC1, RAC3, TRIO,FSCN等也频繁发生了扩增,明确了细胞骨架改变相关通路的变异在转移中的作用,尤其是鉴定了IQGAP3融合作为一个重要的转移相关基因改变,其高水平表达被确定为是不良预后的一个标记物,为肺癌术后复发转移提供了治疗靶点,成果发表于Nat Commun。此外通过对超过100例肺癌标本进行了720个miRNA表达谱研究,发现和鉴定了一批肺癌中的miRNA新靶标,并对其功能及作用机制进行了深入研究(详见第二部分)。
本成果描绘了中国人肺癌尤其肺腺癌的基因位点及表达变异全景图,为肺癌复发转移机制相关分子标志物及靶点的研究奠定了非常重要的基础,也为肺癌的靶向治疗的提供了非常重要且直接指导临床实践的信息。
(2)明确复发转移及耐药机制相关分子标志物及靶点
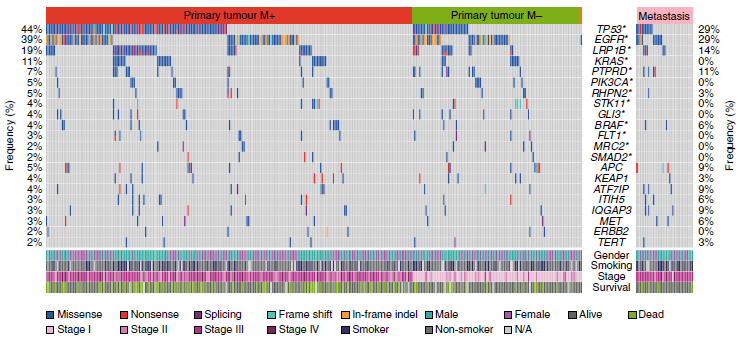
导致肺癌根治手术治疗失败和最终死亡的主要原因是复发或转移,以及对治疗药物耐受等。阐明其分子机制有助于研发针对性的药物和治疗策略,改善患者的总体生存。因此,本实验室在利用标本库获得中国人肺癌特异的基因位点及表达的变异图谱的基础上,针对这些新发现的、与肺癌发生发展密切相关的关键分子进行了功能学及相互作用机制方面的研究,尤其对于可用于预后判断或作为治疗靶点的非编码RNA。如:1)发现miR-497和miR-34a通过共同调节CCNE1基因促进肺癌细胞生长,首次证实miRNA之间存在相互作用并影响癌细胞生长;2)发现miR-26a和-26b通过抑制CDC6的表达,阻抑pre-RC的形成,抑制DNA复制,其在肺癌组织中低表达,与CDC6表达负相关,是肺癌预后独立的不良预测因子,并整合到了后续建立的分子预测模型;3)明确了miR-449a和miR-545抑制肺癌细胞生长、DNA复制及肺癌增殖和侵袭过程的调控机理,与低复发相关;4)lncRNA AF118081在苯并芘诱导支气管细胞恶变中起癌基因样作用,提出lncRNA与化学致癌有关的新概念;5)首次报道MIR-499种子序列变异能显著影响顺铂类药物的临床疗效等等;6)MicroRNA-499 rs3746444T>C变异可导致接受顺铂治疗的肺癌患者生存期缩短,肺癌死亡率增加,细胞试验发现该变异可促进肿瘤细胞生长、增值、转移和侵袭,导致肿瘤细胞耐药,动物试验进一步表明其可减弱顺铂的毒性作用,促进肺肿瘤生长,明确其为接受顺铂治疗的肺癌患者预后的一个功能性生物标志;等。系列成果发表于Cancer Research,Am J Hum Genet,Oncogene,Nucleic Acids Res等权威杂志。
(3)建立和验证了早期非小细胞肺癌根治术后预后判断基因芯片及临床模型
上述成果促使我们将部分成果转化到临床,集中地运用于解决早期肺癌的两大关键问题:通过预后判断的模型解决了患者术后是否需要接受辅助治疗,并探索了复发转移相关的分子靶点,对指导术后具体治疗方案提供了依据。
目前,对肺癌预后的判断及治疗策略的选择主要还是依靠传统的TNM分期来进行评估,但是这一评估方式一直备受争议,尤其是针对早期肺癌患者,相当一部分患者由于风险或预后评估不准确而接受了不必要的“陪绑”化疗。此外,对Ⅰ期非小细胞肺癌尤其是IB期患者在接受肺癌微创根治手术后是否需要接受辅助性的药物治疗在国际上一直存在争论。因此,建立一个准确、简便、无创的肺癌预后预测模型以指导辅助化疗决策是全世界学者的共同期待。本实验室从前述标志物及机制研究的基础上,利用筛选所得标志物,分别建立了:①国际上首个基于14个肺癌预后相关基因的预测芯片,根据每个基因检测结果按对预后影响的权重进行评分并计算每个患者的风险评分,对于非鳞非小细胞肺癌患者来说,随着患者危险度的增加,患者5年生存率降低,根据分值排列将患者分为低危,中危和高危人群,尤其极具争议的Ⅰ期患者。此芯片能有效预测肺癌患者的生存,划分复发风险,指导临床辅助化疗,避免不必要的陪绑药物治疗,并使高危的I期患者得到充分的治疗。其预测作用经中美两国多家中心共同验证,同时适用于东西方人群,相关论文发表于医学顶尖期刊Lancet及JAMA;②构建了基于年龄、性别、病理类型、T分期、N分期、淋巴结采样数目等独立预后因素的nomogram(列线图),其预测的肺癌患者生存期和实际观察到的生存期结果非常吻合,而且预后判别能力明显优于TNM分期系统,并通过国际肺癌专业委员会(IASLC)全球官方数据的验证,提示其广泛适用于NSCLC术后患者,为肺癌患者的预后提供了实用、简便、经济的预后评估工具。相关论文发表于肿瘤学顶级期刊J Clin Oncol;本成果提出的两个预测模型是国际肺癌领域证据级别最高、验证规模最大的,且模型的准确性均得到国际间验证,有直接指导临床的作用,有效地为病人决定辅助治疗与否,避免超过三分一的患者接受陪绑化疗,减少了经济负担。
(三)国内外影响及评价
作为近5年整个肺癌学组研究项目转化成果的分子预测模型,由于其准确性以及东西方人群普适性,相关论文发表于医学顶尖期刊Lancet,JAMA,J Clin Oncol杂志,并多次于世界肺癌大会、欧洲肿瘤学年会等国际会议发言或展示,获国际同行广泛认可:美国德克萨斯西南医学中心的John D Minna在Lancet(Lancet. 2012 Mar 3;379(9818):785-7.)撰文评价该分子预测模型“十分重要,可广泛应用”;加拿大安大略省癌症研究中心的 Paul C. Boutros Boutros在Genome Res(Genome Res. 2015 Oct;25(10):1508-13.)发表评价认为“既往基因生物标记常常不够有效,但此篇论文非常有价值”;中山大学附属第一医院的Jun-Hang Luo在Nat Commun(Nat Commun. 2015 Oct 30;6:8699.)中引述该分子预测模型,并指出:“对比单个生物标记,整合多个生物标志物成一个模型能促进预后价值”;Suresh Senan在Lung Cancer(Lung Cancer. 2016 Apr;94:54-60. )杂志指出“更好的确定和纳入高风险患者进入放化疗,可有效降低死亡率”; 耶鲁大学Thomas Lynch作为嘉宾,在2015年第16届世界肺癌大会的专题评述“Clinicopathologic/Molecular Nomogram”中就此成果进行评述,认为其“很好地运用了生物标志物为临床服务”;此外,美国官方肿瘤研究机构Knight癌症中心将发表于J Clin Oncol的临床模型列为肺癌唯一的预后判断工具,通过网页版推向全球的临床应用。
成果佐证清单
| 序号 | 成果类型 | 成果名称 | 完成人 | 刊物、出版社或授权单位名称 | 年、卷、期、页或专利号 |
|---|---|---|---|---|---|
| 1 | 论文 | A practical molecular assay to predict survival in resected non-squamous, non-small-cell lung cancer: development and international validation studies | Kratz JR, He J, Van Den Eeden SK, Zhu ZH, Gao W, Pham PT, Mulvihill MS, Ziaei F, Zhang H, Su B, Zhi X, Quesenberry CP, Habel LA, Deng Q, Wang Z, Zhou J, Li H, Huang MC, Yeh CC, Segal MR, Ray MR, Jones KD, Raz DJ, Xu Z, Jahan TM, Berryman D, He B, Mann MJ,Jablons DM | Lancet | 2012;379(9818):823-832 |
| 2 | 论文 | Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer | Liang W, Zhang L, Jiang G, Wang Q, Liu L, Liu D, Wang Z, Zhu Z, Deng Q, Xiong X, Shao W, Shi X, He J | Journal of Clinical Oncology | 2015;33(8):861-869 |
| 3 | 论文 | Frequent alterations in cytoskeleton remodelling genes in primary and metastatic lung adenocarcinomas | Wu K, Zhang X, Li F, Xiao D, Hou Y, Zhu S, Liu D, Ye X, Ye M, Yang J, Shao L, Pan H, Lu N, Yu Y, Liu L, Li J, Huang L, Tang H, Deng Q, Zheng Y, Peng L, Liu G, Gu X, He P, Gu Y, Lin W, He H, Xie G, Liang H, An N, Wang H, Teixeira M, Vieira J, Liang W, ZhaoX, Peng Z, Mu F, Zhang X, Xu X, Yang H, Kristiansen K, Wang J, Zhong N, Wang J, Pan-Hammarström Q, He J | Nature Communications | 2015;6:10131 |
| 4 | 论文 | A functional copy-number variation in MAPKAPK2 predicts risk and prognosis of lung cancer | Liu B, Yang L, Huang B, Cheng M, Wang H, Li Y, Huang D, Zheng J, Li Q, Zhang X, Ji W, Zhou Y, Lu J | American Journal of Human Genetics | 2012;91(2):384-390 |
| 5 | 论文 | Sequence Variation in Mature MicroRNA-499 Confers Unfavorable Prognosis of Lung Cancer Patients Treated with Platinum-Based Chemotherapy | Qiu F, Yang L, Ling X, Yang R, Yang X, Zhang L, Fang W, Xie C, Huang D, Zhou Y, Lu J | Clinical Cancer Research | 2015;21(7):1602-1613 |
| 6 | 论文 | MicroRNA-545 suppresses cell proliferation by targeting cyclin D1 and CDK4 in lung cancer cells | Du B, Wang Z, Zhang X, Feng S, Wang G, He J, Zhang B | PLoS One | 2014;9(2):e88022 |
| 7 | 论文 | MicroRNA-192 targeting retinoblastoma 1 inhibits cell proliferation and induces cell apoptosis in lung cancer cells | Feng S, Cong S, Zhang X, Bao X, Wang W, Li H, Wang Z, Wang G, Xu J, Du B, Qu D, Xiong W, Yin M, Ren X, Wang F, He J, Zhang B | Nucleic Acids Research | 2011;39(15):6669-6678 |
| 8 | 论文 | Tumor-suppressive microRNA-449a induces growth arrest and senescence by targeting E2F3 in human lung cancer cells | Ren XS, Yin MH, Zhang X, Wang Z, Feng SP, Wang GX, Luo YJ, Liang PZ, Yang XQ, He JX, Zhang BL | Cancer Letters | 2014;344(2):195-203 |
| 9 | 论文 | Selective killing of lung cancer cells by miRNA-506 molecule through inhibiting NF-κB p65 to evoke reactive oxygen species generation and p53 activation | Yin M, Ren X, Zhang X, Luo Y, Wang G, Huang K, Feng S, Bao X, Huang K, He X, Liang P, Wang Z, Tang H, He J, Zhang B | Oncogene | 2015;34(6):691-703 |
| 10 | 论文 | The p53-induced lincRNA-p21 derails somatic cell reprogramming by sustaining H3K9me3 and CpG methylation at pluripotency gene promoters | Bao X, Wu H, Zhu X, Guo X, Hutchins AP, Luo Z, Song H, Chen Y, Lai K, Yin M, Xu L, Zhou L, Chen J, Wang D, Qin B, Frampton J, Tse HF, Pei D, Wang H, Zhang B, | Cell Research | 2015;25(1):80-92 |
| 11 | 论文 | MicroRNA-26a/b regulate DNA replication licensing, tumorigenesis, and prognosis by targeting CDC6 in lung cancer | Zhang X, Xiao D, Wang Z, Zou Y, Huang L, Lin W, Deng Q, Pan H, Zhou J, Liang C, He J | Molecular Cancer Research | 2014;12(11):1535-1546 |
| 12 | 论文 | Oncogenic role of long noncoding RNA AF118081 in anti-benzo[a]pyrene-trans-7,8-dihydrodiol-9,10-epoxide-transformed 16HBE cells | Yang Q, Zhang S, Liu H, Wu J, Xu E, Peng B, Jiang Y | Toxicology Letters | 2014;229(3):430-439 |
| 13 | 论文 | Sequence Variation in Mature MicroRNA-499 Confers Unfavorable Prognosis of Lung Cancer Patients Treated with Platinum-Based Chemotherapy | Qiu F, Yang L, Ling X, Yang R, Yang X, Zhang L, Fang W, Xie C, Huang D, Zhou Y, Lu J | Clinical Cancer Research | 2015;21(7):1602-1613 |
| 14 | 论文 | Oncogenic driver mutations in patients with non-small-cell lung cancer at various clinical stages | Zhou JX, Yang H, Deng Q, Gu X, He P, Lin Y, Zhao M, Jiang J, Chen H, Lin Y, Yin W, Mo L, He J | Annals of Oncology | 2013;24(5):1319-1325 |
| 15 | 论文 | Selective killing of lung cancer cells using carcinoembryonic antigen promoter and double suicide genes, thymidine kinase and cytosine deaminase (pCEA-TK/CD) | Qiu Y, Peng GL, Liu QC, Li FL, Zou XS, He JX | Cancer Letters | 2012;316(1):31-38. |